生命現象を支える生体高分子は、「立体構造」とその水中での揺らぎの様子「運動性」の両方の性質を持っている。立体構造は分子認識や酵素活性など個々のタンパク質が担う機能の基盤であり、運動性はその機能を厳密に制御していることが知られている。
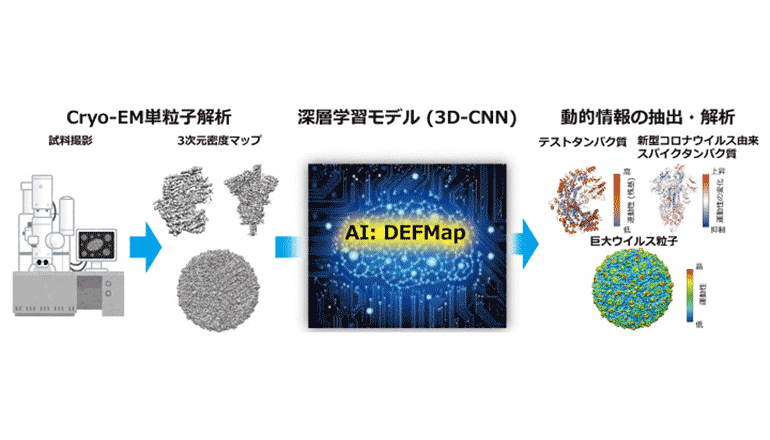
病態解明や医薬品開発で必要不可欠となる。タンパク質の運動性を解析するには、専門的な実験計測や科学演算などが必要であり、容易ではない。近年発展が著しいクライオ電子顕微鏡(cryo-EM)は、単粒子解析を通じて、上記立体構造を原子~近原子分解能で次々と明らかにしているが、解析対象の生体高分子は一般に巨大かつ複雑であるため、従来、それらの運動性情報(柔らかさ)を得ることは技術的に困難だったという。
理研MIH医薬プロセス最適化プラットフォーム推進グループ、京都大学大学院医学研究科、横浜市立大学大学院生命医科学研究科、大阪大学蛋白質研究所蛋白質構造生物学研究部門――4者の共同研究グループは、AI技術の一つ深層学習(3D-CNN)と分子動力学(MD)計算を組み合わせることで、cryo-EMで計測される立体構造データのみから、タンパク質の運動性情報を直接抽出する新たな手法の開発に成功した。
原子レベルで簡便・迅速に推定できる画期的なAI「DEFMap」の学習済みモデルをGitHub上に公開。あらゆる研究者が3次元密度マップから簡便に運動性を抽出・確認できる。これにより、これまで解析が困難であった生体高分子の運動性が明らかになり、機能メカニズムに対する新たな知見の発見につながると考えられる。同手法は超巨大なウイルス粒子などにも適用可能だという。
タンパク質やDNAなどの生体高分子の運動性を解析する新たなアプローチを提供し、生命科学の進展や医薬品開発への貢献が期待される。研究グループの成果は『Nature Machine Intelligence』電子版に掲載された。
