山本量一 工学研究科教授、John Molina 同助教、Simon Schnyder 福井謙一記念研究センター特定研究員らの研究グループらが成功。研究成果は、2017年7月12日に英国の科学誌「Scientific Reports」に掲載された。
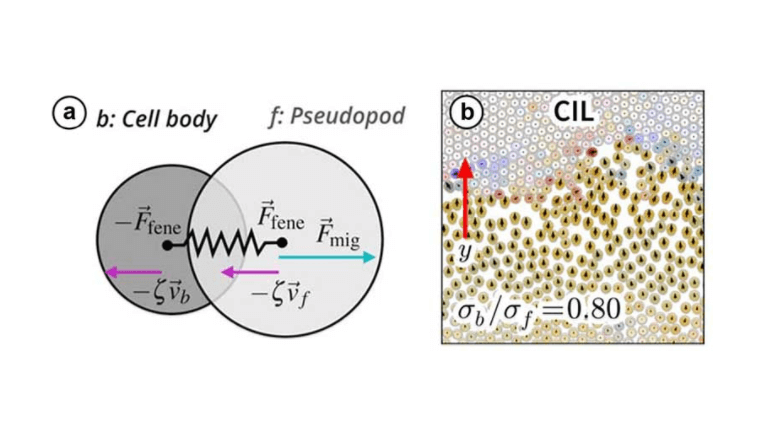
研究グループは、実際の細胞集団が示す種々の複雑な集団運動を再現することができる、簡単な細胞モデルの構築に成功。このモデルでは、基板上を遊走する細胞をバネでつながった二つの円盤(仮足部分を表す前部の円盤と細胞本体を表す後部の円盤が細胞骨格で結合されたもの)として表現し、円盤間の距離で示される細胞の伸びに依存した推進力を導入することで、接触阻害の効果を取り入れている。
■図:(a) 自走する細胞をバネでつながった二つの円盤でモデル化し、細胞の伸び(円盤間の距離)に依存した推進力を導入することで接触阻害(Contact inhibition of locomotion、CIL)を取り入れる
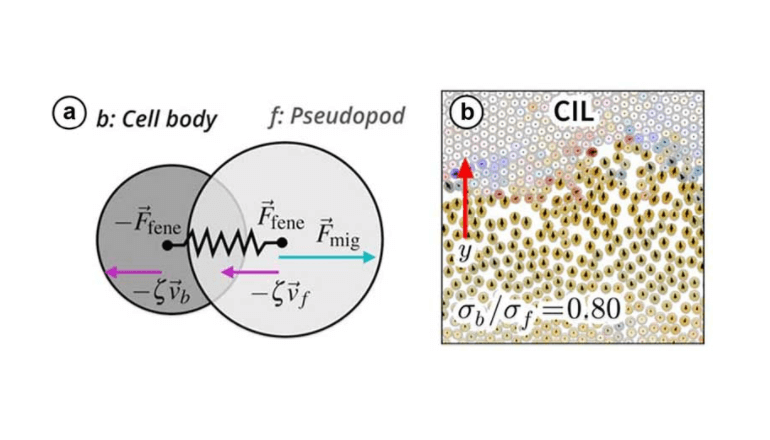
(b) シミュレーション結果の一例。上部の混雑した渋滞領域に向かって自走する細胞の集団運動の再現に成功した
同モデルでは、細胞が他の細胞と接触すると減速すること、基板上の細胞数密度の増加とともに不動状態に陥ることを正しく再現。細胞の形が集団運動に大きな影響を与えることを発見した。
計算科学の導入は始まっている生物科学の分野だが、これまでの成功例の多くは細胞内のDNAや蛋白質など、ごく一部の生体分子を対象としたものに限られていた。多数の細胞からなる生体組織を対象とした場合、関与する分子が膨大な数に上り、細胞分裂などの非常に遅い現象をも考慮する必要があった。そのため、既存の分子モデルをそのまま適用するのは非常に難しかったという。
研究グループは「粗視化モデリング」と呼ばれる手法を発展させ、生体組織に対して有効なシミュレーション手法の構築を目指している。研究者の1人である山本氏は「最も顕著な研究成果は、膨大な数の分子からなる細胞に対して簡単な物理モデルを提案し、生体組織内部で見られる細胞の複雑な集団運動が、実は非常に単純なメカニズムで起こり得ることを示したことにある」とコメントしている。