理研計算科学研究機構粒子系生物物理研究チームの松永康佑研究員(JSTさきがけ研究員)、横浜市立大学大学院の山根努特任助教、池口満徳教授、木寺詔紀教授、東京工業大学の村上聡教授らの共同研究グループが成功。研究成果は、英国のオンライン科学雑誌『eLife』に掲載された。
効くはずの薬が効かなくなるという「薬剤耐性化」の問題は、現代の医療現場で大きな問題となっている。病原菌やがん細胞がこのような薬剤耐性を持つメカニズムは幾つかあるが、細胞膜に埋まっている多剤排出トランスポーターと呼ばれるタンパク質が原因の一つであることが分かっている。
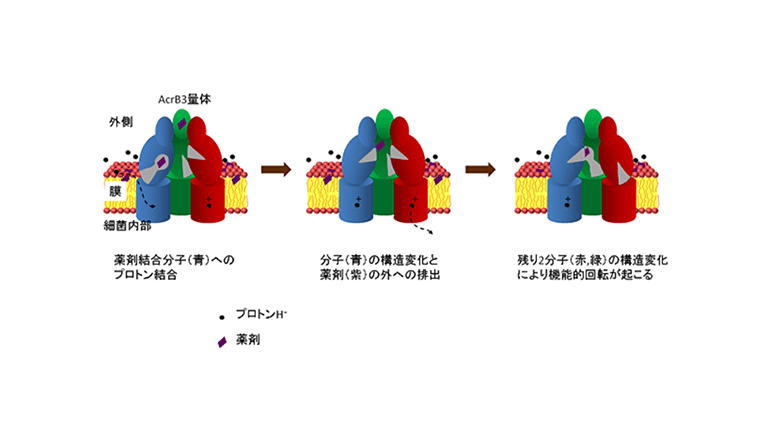
大腸菌由来の多剤排出トランスポーターであるAcrBは、村上教授らが2002年および2006年にX線結晶構造解析によって構造を解明し、それに基づいた作動原理として「機能的回転機構」仮説を提唱していた。
2010年には、この仮説は京都大学の高田彰二教授らによる粗視化シミュレーション技法などによって実証されまが、膜を介したプロトン(水素イオン)移動によってどのようにAcrBの構造変化が起き、機能的回転が起るのか、その動的な機構は分かっていなかった。
今回、共同研究グループは、AcrBとその周りの膜や水分子を、丸ごと模した全原子モデルを計算機上で再現し、京の高並列性を生かすアルゴリズムを用いて薬剤排出過程を計算。その結果、AcrBの膜内にあるアスパラギン酸にプロトンが結合すると、薬剤排出へつながる構造変化が引き起こされること、また50オングストローム(Å、1Åは100億分の1メートル)も離れた薬剤排出部位の構造変化へ至る過程を明らかにした。
多くの生体分子にみられるプロトン結合による化学エネルギーと薬剤輸送という力学的エネルギーの間の変換という基礎的問題に対して、一つの機構を提供する。また、多剤排出トランスポーターによって排出されない薬剤や、排出を阻害する薬剤の開発に資すると期待できるという。
